杰毅生物荣膺 2024 年度杭州首批 “新雏鹰企业”
近日,杭州市科学技术局正式公布 2024 年度杭州市 “新雏鹰企业” 认定名单。经过层层筛选与严格评审,拟认定杭州市 “新雏鹰” 企业 102 家。杭州杰毅生物技术有限公司凭借在感染性疾病分子诊断领域展现出的技术创新能力、市场表现与潜力等方面荣膺首批 “新雏鹰企业” 称号!

新雏鹰计划——培育未来产业新锐力量
“新雏鹰企业” 由杭州市科学技术局组织实施,为培育未来产业新动能而设立的专项培育计划,瞄准的是杭州市重点布局的未来产业。“新雏鹰计划” 的入选标准涵盖企业的技术创新能力、研发投入水平、知识产权成果、产业化前景和市场竞争力,旨在筛选和扶持前沿科技领域展现出强劲创新能力、高成长潜力、产业化前景的科技型企业。2024 年度全市仅 102 家企业获批,它们将成为杭州市构建现代化产业体系、打造创新创业新天堂的重要生力军。
杰毅生物——创新驱动感染精准诊疗
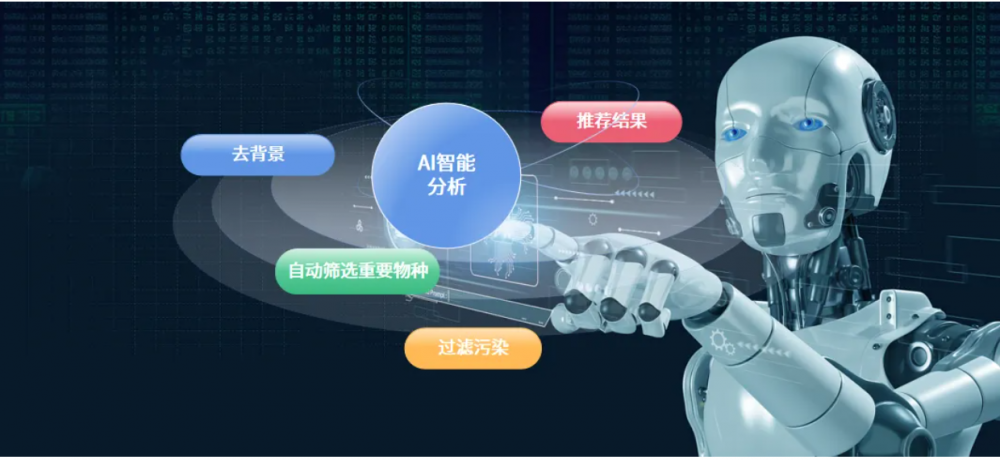
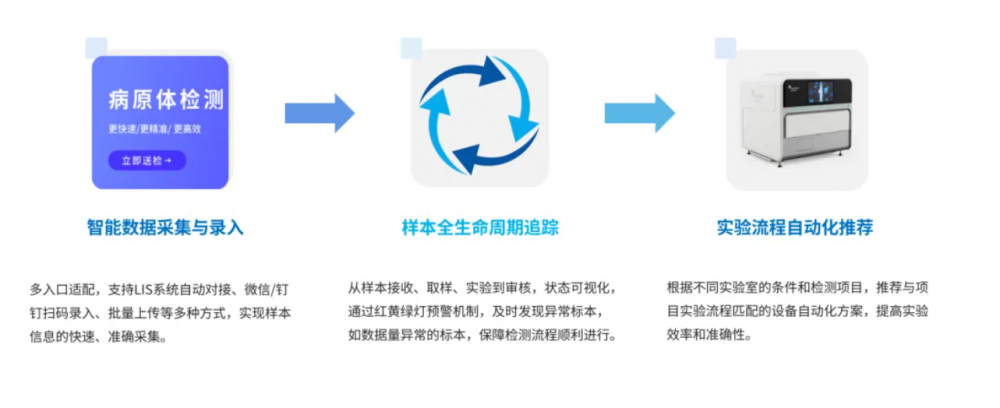
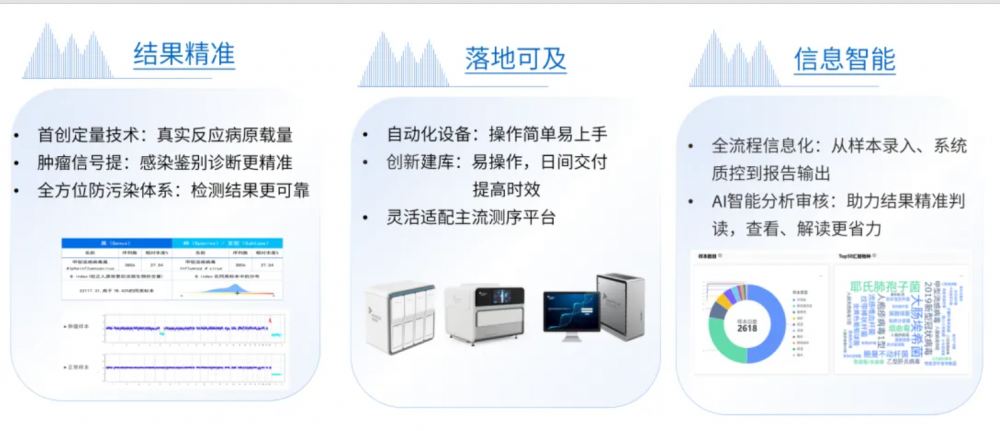
杰毅生物深耕感染性疾病分子诊断领域,将数据驱动能力深度渗透至分子诊断产品开发与应用的全链路,成功为客户打造出集自动化实验流程、数字化实验室管理、智能化报告分析于一体的一体化诊断解决方案。凭借在病原 NGS 领域的技术产品开发优势与丰富的实践经验,杰毅生物连续三年以优异成绩通过国家卫健委临检中心发起的 mNGS 室间质评,充分印证了公司高度标准化的 mNGS 检测能力与稳定成熟的实验室管理水平。

针对不同规模客户及多样化场景的 NGS 检测需求,公司于 2025 年正式推出覆盖泛感染、呼吸、中枢、生殖、分枝杆菌等症候群 tNGS 系列产品,并为本地化开展病原 NGS 检测的客户带来 “毅达 Lab” 病原体检测整体解决方案,具备实验流程更简单高效、检测结果更精准、成本更集约的优势,帮助更多医疗机构实现本地化优质高效的病原 NGS 检测,进一步释放技术产业化和市场拓展中的巨大潜力。

随着此次入选 “新雏鹰” 企业,杰毅生物将获得杭州市政府在政策指导、研发补助等持续赋能,公司也将加大前沿技术产品开发和市场拓展力度,为广大医疗卫生机构带来更精准、更智慧的病原诊断解决方案,加速成为引领感染性疾病精准诊断创新发展的生力军。